Phương pháp resampling là một kỹ thuật thống kê quan trọng, cho phép ước lượng độ không chắc chắn của một ước lượng thống kê hoặc kiểm tra giả thuyết thông qua việc lặp lại chọn mẫu dữ liệu. Điều này thực hiện bằng cách tạo ra nhiều mẫu dữ liệu giả từ mẫu dữ liệu gốc và tiến hành phân tích trên các mẫu giả này để thu được phân phối của ước lượng. Trong thế giới dữ liệu ngày nay, khi lượng dữ liệu tăng lên nhanh chóng và đôi khi chất lượng dữ liệu không đảm bảo, resampling trở thành công cụ không thể thiếu giúp nhà khoa học dữ liệu và nhà nghiên cứu có được cái nhìn sâu sắc hơn về dữ liệu và đưa ra các quyết định dựa trên dữ liệu có cơ sở vững chắc hơn.
Cơ sở lý thuyết của phương pháp resampling
Cơ sở lý thuyết của phương pháp resampling dựa trên ý tưởng rằng các mẫu tái chọn (resample) từ dữ liệu gốc có thể cung cấp thông tin quý giá về phân phối của ước lượng thống kê hoặc giúp kiểm tra các giả thuyết thống kê mà không cần dựa vào giả định phức tạp về dạng phân phối của dữ liệu. Nguyên tắc hoạt động của resampling là tạo ra một số lượng lớn mẫu tái chọn từ dữ liệu gốc, và từ đó, tính toán ước lượng thống kê hoặc thực hiện kiểm định giả thuyết trên mỗi mẫu tái chọn này. Kỹ thuật này cho phép ước lượng độ không chắc chắn của ước lượng thống kê hoặc xác định sự khác biệt có ý nghĩa thống kê giữa các nhóm mà không cần giả định về phân phối của dữ liệu.
So với các phương pháp truyền thống, resampling mang lại một số lợi ích đáng kể:
- Không yêu cầu giả định về phân phối: Không giống như nhiều phương pháp thống kê cổ điển đòi hỏi dữ liệu phải tuân theo một phân phối cụ thể (ví dụ, phân phối chuẩn), resampling không yêu cầu giả định này, làm cho nó trở nên linh hoạt và áp dụng được cho nhiều loại dữ liệu khác nhau.
- Dễ dàng ước lượng độ không chắc chắn: Resampling cung cấp một phương pháp trực tiếp để ước lượng khoảng tin cậy và độ không chắc chắn của ước lượng thống kê, qua đó giúp ra quyết định dựa trên dữ liệu một cách chính xác hơn.
- Phù hợp với dữ liệu lớn và phức tạp: Trong thời đại dữ liệu lớn, resampling phát huy tác dụng mạnh mẽ trong việc phân tích dữ liệu phức tạp, cho phép nhà nghiên cứu khám phá dữ liệu và mô hình hóa mà không bị hạn chế bởi giả định truyền thống.
Ứng dụng của resampling trong thực tiễn rất đa dạng, bao gồm:
- Ước lượng khoảng tin cậy: Sử dụng phương pháp Bootstrap để ước lượng khoảng tin cậy cho trung bình, trung vị, phương sai, và các ước lượng thống kê khác.
- Kiểm định giả thuyết: Áp dụng kiểm định Permutation để so sánh sự khác biệt giữa các nhóm, mà không cần giả định về phân phối của dữ liệu.
- Mô phỏng và phân tích rủi ro: Trong tài chính và kinh doanh, resampling được sử dụng để mô phỏng các kịch bản khác nhau và ước lượng rủi ro.
Nhờ vào sự linh hoạt và hiệu quả, phương pháp resampling ngày càng trở nên phổ biến trong nghiên cứu khoa học dữ liệu, đóng vai trò là công cụ mạnh mẽ giúp nhà nghiên cứu đưa ra những phát hi
Monte Carlo
Các phương pháp Monte Carlo thường được sử dụng trong các mô phỏng trong đó phân phối mẹ được biết đến hoặc được giả định. Nói không ngoa, tôi đã sử dụng phương pháp Monte Carlo trong suốt học kỳ để minh họa cách thức hoạt động của các kỹ thuật thống kê.
Ví dụ: giả sử chúng ta muốn mô phỏng phân phối của thống kê F cho hai mẫu được rút ra từ phân phối chuẩn. Chúng tôi sẽ chỉ định giá trị trung bình và phương sai của phân phối chuẩn, tạo hai mẫu có kích thước nhất định và tính toán tỷ lệ phương sai của chúng. Chúng tôi sẽ lặp lại quá trình này nhiều lần để tạo ra phân phối tần số của thống kê F, từ đó chúng tôi có thể tính toán giá trị p hoặc giá trị tới hạn.
Đây là một ví dụ về Monte Carlo trong R. Khối mã đầu tiên tạo ra một hàm tính tỷ lệ F của hai mẫu, với kích thước mẫu n1 và n2. Khối mã thứ hai khai báo các kích thước mẫu mà chúng ta muốn mô phỏng, sau đó gọi hàm của chúng ta nhiều lần để xây dựng phân phối các giá trị F.
simulateOneF <- function(n1, n2) {
data1 <- rnorm(n1)
data2 <- rnorm(n2)
F <- var(data1) / var(data2)
F
}
n1 <- 12
n2 <- 25
F <- replicate(10000, simulateOneF(n1, n2))Chúng ta có thể tính toán giá trị p bằng cách tìm phần trăm các giá trị F của chúng ta bằng hoặc vượt quá một thống kê F quan sát được (trong ví dụ này là 3,5):
Fobserved <- 3.5 pValue <- length(F[F >= Fobserved]) / length(F) pValue
Điều này cho thấy tỷ lệ giá trị F vượt quá mức quan sát của F là 3,5, vì vậy nó là giá trị p một phía. Để nhận được giá trị p hai phía, hãy nhân đôi xác suất này.
Vì giá trị p có thể được tính như thế này cho phân phối dự kiến của bất kỳ thống kê nào, chúng ta có thể viết hàm tính giá trị p cho thống kê cho bất kỳ phân phối nào được tạo bằng phương pháp lấy mẫu lại như Monte Carlo:
pvalue <- function(x, observed, tails=1) {
nExtreme <- length(x[ x>= observed])
nTotal <- length(x)
p <- nExtreme / nTotal * tails
p
}Gọi điều này rất đơn giản: đối số đầu tiên là phân phối được cung cấp bởi Monte Carlo (hoặc một phương pháp lấy mẫu lại khác) và đối số thứ hai là thống kê được quan sát. Theo mặc định, nó sẽ tính giá trị p một bên (bên phải); chỉ định đuôi = 2 cho giá trị p hai đuôi. Việc gọi một hàm tự tạo như thế này tạo ra mã dễ đọc hơn và dễ hiểu hơn:
pvalue(F, Fobserved)
Tóm lại, có ba bước để có một Monte Carlo:
- mô phỏng các quan sát dựa trên phân phối lý thuyết
- nhiều lần tính toán thống kê quan tâm để xây dựng phân phối của nó dựa trên giả thuyết rỗng
- sử dụng phân phối của thống kê để tìm giá trị p hoặc giá trị tới hạn.
Randomization
Randomization là một phương pháp lấy lại mẫu đơn giản và mô hình suy nghĩ minh họa cho cách tiếp cận. Giả sử chúng ta có hai mẫu A và B, trên đó chúng ta đã đo lường một thống kê (ví dụ: giá trị trung bình). Một cách để hỏi liệu hai mẫu có phương tiện khác nhau đáng kể hay không là hỏi khả năng chúng ta quan sát thấy sự khác biệt về phương tiện của chúng như thế nào nếu các quan sát được phân bổ ngẫu nhiên cho hai nhóm. Chúng tôi có thể mô phỏng điều đó bằng cách chỉ định ngẫu nhiên từng quan sát cho nhóm A hoặc B, tính toán sự khác biệt của các phương tiện và lặp lại quá trình này cho đến khi chúng tôi xây dựng được phân phối của sự khác biệt về phương tiện. Bằng cách làm này, chúng tôi đang xây dựng sự phân bố của sự khác biệt về phương tiện theo giả thuyết rỗng rằng hai mẫu đến từ một tập hợp.
Từ phân phối này, chúng tôi có thể tìm thấy các giá trị quan trọng mà chúng tôi có thể so sánh sự khác biệt quan sát được về giá trị trung bình. Chúng tôi cũng có thể sử dụng phân phối này để tính toán giá trị p của thống kê quan sát.
Các bước thiết yếu trong Randomization là:
- xáo trộn các quan sát giữa các nhóm
- tính toán nhiều lần các thống kê quan tâm
- sử dụng phân phối đó của thống kê để tìm các giá trị quan trọng hoặc giá trị p
Đây là một ví dụ về việc thực hiện một phép ngẫu nhiên để đánh giá sự khác biệt về phương tiện của hai mẫu.
# The data
x <- c(0.36, 1.07, 1.81, 1.67, 1.16, 0.18, 1.26, 0.61, 0.08, 1.29)
y <- c(1.34, 0.32, 0.34, 0.29, 0.64, 0.20, 0.50, 0.85, 0.38, 0.31, 2.50, 0.69, 0.41, 1.73, 0.62)
# Function for calculating one randomized difference in means
randomizedDifferenceInMeans <- function(x, y) {
nx <- length(x)
ny <- length(y)
# combine the data
combined <- c(x, y)
ncom <- length(combined)
indices <- 1:ncom
# initially assign all observations to y group
group <- rep('y', ncom)
# assign a subsample to to x group
xsub <- sample(indices, nx)
group[xsub] <- 'x'
# calculate the means
meanX <- mean(combined[group=='x'])
meanY <- mean(combined[group=='y'])
differenceInMeans <- meanX - meanY
differenceInMeans
}
# Repeat that function many times to build the distribution
diffMeans <- replicate(10000, randomizedDifferenceInMeans(x, y))Tại thời điểm này, chúng tôi có thể làm một số điều. Bởi vì chúng tôi có một phân phối cho giả thuyết rỗng, chúng tôi có thể so sánh sự khác biệt quan sát được về giá trị trung bình với các giá trị tới hạn hai phía. Từ đó, chúng ta có thể chấp nhận hoặc bác bỏ giả thuyết không có sự khác biệt về phương tiện. Hàm quantile () giúp việc tìm kiếm các giá trị tới hạn trở nên dễ dàng.
observedDifference <- mean(x) - mean(y) alpha <- 0.05 quantile(diffMeans, alpha/2) quantile(diffMeans, 1-alpha/2)
Đôi khi, sẽ hữu ích nếu hiển thị phân phối với các giá trị tới hạn và giá trị quan sát. Điều này cho phép người đọc hiểu hình dạng của phân phối và hình dung giá trị quan sát đã giảm so với các giá trị tới hạn ở đâu.
hist(diffMeans, breaks=50, col='gray', xlab='Difference of Means', main='') observedDifference <- mean(x) - mean(y) abline(v=observedDifference, lwd=4, col='black') alpha <- 0.05 abline(v=quantile(diffMeans, alpha/2), col='red', lwd=2) abline(v=quantile(diffMeans, 1-alpha/2), col='red', lwd=2)
Thay vì các giá trị tới hạn, chúng ta có thể tính toán giá trị p hai bên cho sự khác biệt quan sát được về phương tiện, bằng cách sử dụng hàm giá trị p mà chúng ta đã tạo ở trên.
observedDifference <- mean(x) - mean(y) pvalue(diffMeans, observedDifference, tails=2)
Bootstrap
Bootstrap là một kỹ thuật mạnh mẽ, linh hoạt và trực quan để xây dựng khoảng tin cậy. Cái tên bootstrap xuất phát từ ý tưởng “nâng cao bản thân bằng chính bootstraps của bạn”: nó xây dựng các giới hạn tin cậy từ việc phân phối dữ liệu. Giả định chính là dữ liệu là một mô tả tốt về sự phân bố tần suất của dân số và sẽ đúng nếu bạn thu thập một mẫu ngẫu nhiên bằng cách tuân theo thiết kế lấy mẫu tốt.
Nguyên tắc đằng sau bootstrap là rút ra một tập hợp các quan sát từ dữ liệu để tạo ra một mẫu mô phỏng có cùng kích thước với mẫu ban đầu. Việc lấy mẫu này được thực hiện cùng với việc thay thế, có nghĩa là một quan sát cụ thể có thể được lấy mẫu ngẫu nhiên nhiều lần trong một thử nghiệm cụ thể, trong khi những quan sát khác có thể hoàn toàn không được lấy mẫu. Thống kê được tính toán trên mẫu khởi động này và quá trình này được lặp lại nhiều lần (hàng nghìn lần trở lên) để xây dựng phân phối cho thống kê. Giá trị trung bình của các giá trị khởi động này là ước tính của tham số.
Việc phân phối các giá trị khởi động được sử dụng để xây dựng khoảng tin cậy trên ước tính tham số. Để làm điều này, các giá trị khởi động được sắp xếp từ thấp nhất đến cao nhất. Phần tử 1-α / 2% (ví dụ: 0,025%) và phần tử α / 2% (ví dụ: 0,975%) tương ứng với các giới hạn tin cậy dưới và trên. Ví dụ: nếu chúng tôi tính toán thống kê của mình 10.000 lần và muốn có giới hạn tin cậy 95%, chúng tôi sẽ tìm thấy các phần tử ở hàng 250 và 9750 của vectơ đã sắp xếp của thống kê khởi động. Hàm quantile () thực hiện tất cả những điều này một cách tự động, giúp chúng ta không gặp rắc rối về xác định và tìm điểm cắt.
Độ tin cậy của bootstrap được cải thiện với số lượng bản sao bootstrap. Việc tăng số lượng bản sao sẽ làm tăng thời gian tính toán, nhưng đây hiếm khi là vấn đề như trước đây.
Bootstrap một biến
Ví dụ này minh họa một sơ đồ khởi động của độ lệch chuẩn mẫu. Điều này có thể được sửa đổi dễ dàng đối với bất kỳ thống kê nào được tính toán trên một biến duy nhất. Bước đầu tiên là xây dựng một hàm tính toán một thống kê khởi động từ dữ liệu. Tiếp theo, hàm replicate () được sử dụng để gọi hàm này nhiều lần trên dữ liệu của chúng tôi để xây dựng phân phối thống kê khởi động.
bootstrapSd <- function(x) {
bootstrappedSample <- sample(x, size=length(x), replace=TRUE)
theSd <- sd(bootstrappedSample)
theSd
}
x <- c(1.61, 0.84, 0.01, 0.35, 0.37, 1.13, 0.25, 1.25, 3.24, 0.68)
sd <- replicate(10000, bootstrapSd(x))Giá trị trung bình của phân phối các mẫu khởi động này là ước tính của chúng tôi về tham số. Sử dụng quantile () và mức độ tin cậy của bạn (alpha) để tính khoảng tin cậy trên ước tính tham số đó. Độ chính xác của những ước tính này có thể được tăng lên bằng cách thực hiện một số lượng lớn hơn các bản sao, nhưng với chi phí tăng thời gian tính toán.
parameterEstimate <- mean(sd) alpha <- 0.05 lowerCL <- quantile(sd, alpha/2) upperCL <- quantile(sd, 1-alpha/2)
Bootstrap hai biến
Bootstrap cũng có thể được sử dụng trên một thống kê dựa trên hai biến, chẳng hạn như hệ số tương quan hoặc độ dốc. Điều này chỉ phức tạp hơn một chút so với ví dụ trước, vì chúng ta cần lấy mẫu các hàng dữ liệu, nghĩa là, vẽ ngẫu nhiên cả hai biến tương ứng với một điểm dữ liệu hoặc quan sát duy nhất. Chọn độc lập một phần tử cho mỗi biến là một sai lầm phổ biến khi xây dựng khoảng tin cậy, vì nó phá hủy cấu trúc tương quan trong tập dữ liệu. Người ta có thể làm điều này một cách có chủ ý, điều này sẽ xây dựng một phân phối cho giả thuyết rỗng (nghĩa là các giá trị không tương quan) và điều này có thể được sử dụng để tính toán các giá trị quan trọng hoặc giá trị p. Cách tiếp cận phổ biến hơn nhiều là phương pháp hiển thị bên dưới, vì nó được sử dụng để ước tính tham số và khoảng tin cậy trên chúng.
bootstrapR <- function(x, y) {
i <- 1:length(x)
theSample <- (sample(i, length(x), replace=TRUE))
pearsonR <- cor(x[theSample], y[theSample])
pearsonR
}
x <- c(3.37, 2.04, 0.67, 0.99, 0.91, 2.25, 5.54, 2.67, 2.43, 2.00)
y <- c(12.30, 9.91, 5.92, 8.56, 6.51, 10.53, 19.22, 13.52, 10.47, 9.03)
r <- replicate(10000, bootstrapR(x, y))Một bootstrap hai biến được minh họa ở đây cho một hệ số tương quan. Như trước đây, bước đầu tiên là viết một hàm tính toán một giá trị khởi động duy nhất của hệ số tương quan. Trong hàm này, sample () được sử dụng để vẽ một mẫu có cùng kích thước với mẫu ban đầu, nhưng với một tập hợp các quan sát được chọn ngẫu nhiên từ dữ liệu. Đảm bảo đặt Replace = TRUE nếu không điều này sẽ luôn tạo ra chính xác tập dữ liệu ban đầu, khiến tất cả các giá trị khởi động giống hệt nhau. Sau hàm, sử dụng replicate () để xây dựng phân phối thống kê khởi động.
parameterEstimate <- mean(r) alpha <- 0.05 quantile(r, alpha/2) quantile(r, 1-alpha/2)
Ước tính tham số và khoảng tin cậy của nó được tìm thấy theo cùng một cách với bootstrap một biến.
Bootstrap đa biến
Cách tiếp cận hai biến có thể được tổng quát hóa cho dữ liệu đa biến. Để xây dựng khoảng tin cậy, hãy chọn ngẫu nhiên một hàng của khung dữ liệu như được thực hiện trong trường hợp hai biến và sử dụng tất cả các quan sát cho trường hợp đó khi mẫu khởi động được tạo. Nếu muốn các giá trị p và giá trị tới hạn, hãy chọn ngẫu nhiên các quan sát của từng biến, nghĩa là các biến không nhất thiết phải từ cùng một hàng của khung dữ liệu.
Một số phương pháp (chẳng hạn như chia tỷ lệ đa chiều không theo hệ mét) không cho phép các trường hợp giống hệt nhau và điều này sẽ gây ra sự cố cho một bootstrap đa biến. Một giải pháp phổ biến là thêm một số ngẫu nhiên rất nhỏ vào mỗi giá trị khi trường hợp đó được chọn và điều này sẽ ngăn ngừa vấn đề về các trường hợp giống hệt nhau. Ví dụ: nếu các giá trị dữ liệu nằm trong khoảng từ 1–10, thì số ngẫu nhiên có thể ở thang điểm 0,001; điều này đủ để ngăn các giá trị không giống nhau, nhưng không đủ để thay đổi cơ bản dữ liệu hoặc kết quả.
Jackknife
Jackknife là một kỹ thuật khác để ước tính một tham số và đặt giới hạn tin cậy cho nó. Tên đề cập đến việc cắt dữ liệu, bởi vì tôi hoạt động bằng cách loại bỏ một quan sát duy nhất, tính toán thống kê mà không có một giá trị đó, sau đó lặp lại quá trình đó cho mỗi quan sát (chỉ loại bỏ một giá trị, tính toán thống kê). Điều này tạo ra một phân phối các giá trị jackknifed của thống kê của chúng tôi, từ đó ước tính tham số và khoảng tin cậy được tính toán.
Cụ thể, giả sử chúng ta quan tâm đến tham số K, nhưng chúng ta chỉ có một ước lượng về nó (thống kê k) dựa trên mẫu n quan sát của chúng ta.
Để tạo mẫu jackknifed đầu tiên bao gồm n-1 quan sát, hãy loại bỏ quan sát đầu tiên trong dữ liệu. Tính toán thống kê (được gọi là k-i) trên mẫu đó, trong đó chỉ số con -i có nghĩa là tất cả các quan sát ngoại trừ thứ i. Từ thống kê này, hãy tính một đại lượng được gọi là giá trị giả κi:

Lặp lại quá trình này bằng cách thay thế lần quan sát đầu tiên và loại bỏ lần quan sát thứ hai, để tạo một mẫu mới có kích thước n-1. Tính toán thống kê và giá trị giả như trước đây. Lặp lại điều này cho mọi quan sát có thể cho đến khi có n giá trị giả, mỗi giá trị có cỡ mẫu n-1. Từ n giá trị giả này, chúng ta có thể ước tính tham số, sai số chuẩn của nó và khoảng tin cậy.
Ước tính jackknifed của tham số K bằng giá trị trung bình của các giá trị giả:

Sai số chuẩn của K bằng độ lệch chuẩn của các giá trị psuedoval chia cho căn bậc hai của cỡ mẫu (n):
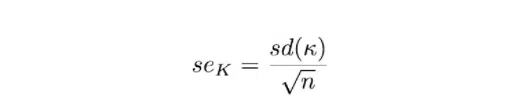
Giới hạn tin cậy jackknifed dựa trên ước tính của K, sai số chuẩn và điểm t tới hạn hai phía với n-1 bậc tự do:

Có hai lưu ý cho con dao. Đầu tiên, khoảng tin cậy giả định rằng thống kê jackknifed được phân phối chuẩn, được chứng minh bằng định lý giới hạn trung tâm cho các mẫu lớn. Nếu kích thước mẫu của chúng ta nhỏ, định lý giới hạn trung tâm có thể không đảm bảo tính chuẩn. Thứ hai, các giá trị giả không độc lập, chúng nhất thiết phải tương quan.
Đây là một minh chứng về một con dao nhỏ, trong đó số liệu thống kê mà chúng tôi muốn con dao bấm là độ lệch chuẩn. Mặc dù không cần thiết đối với một vấn đề đơn giản như thế này, nhưng tôi đặt nó bên trong hàm allowStatistic () để nhấn mạnh rằng nếu chúng ta muốn tạo ra một thống kê khác, tất cả những gì chúng ta phải làm là thay đổi nội dung của CalculStatistic () và phần còn lại của mã sẽ không thay đổi.
calculateStatistic <- function(x) {
sd(x)
}x <- c(0.12, 0.97, 1.03, 0.10, 0.20, 0.22, 0.63, 0.21, 2.36, 3.07, 0.91, 0.65, 0.71, 0.13, 2.37, 1.44, 0.55, 1.31, 0.29, 0.25, 2.32, 1.84, 1.03, 0.42, 0.34, 0.37, 0.02, 1.46, 0.62, 0.73, 1.01, 0.37, 2.23, 0.10, 0.53, 0.31, 0.38, 0.20, 5.12, 2.02, 2.35, 0.07, 0.03, 0.13, 2.24, 0.70, 0.19, 1.07, 1.88, 2.61)
Tiếp theo, tính toán các giá trị giả. Đầu tiên, tạo một vectơ để chứa các giá trị giả, tính toán thống kê (stat) trên tất cả dữ liệu. Tiếp theo, lặp lại dữ liệu, loại bỏ từng quan sát một. Vòng lặp for này sẽ thực hiện lặp đi lặp lại tất cả các thao tác bên trong dấu ngoặc nhọn. Đầu tiên, tập dữ liệu jackknifed được trích xuất (jack), sau đó thống kê được tính toán trên nó (jackStat), sau đó giá trị giả được lưu vào vector mà chúng tôi đã tạo. Trong lần đầu tiên vượt qua vòng lặp, tôi sẽ bằng 1 và nó sẽ được tăng thêm 1 trên mỗi lần vượt qua. Trên đường chuyền cuối cùng, nó sẽ có độ dài bằng (x), tức là kích thước mẫu của chúng tôi. Mỗi giá trị giả của chúng ta được lưu vào vectơ mà chúng ta đã tạo ở bước cuối cùng trong vòng lặp.
pseudovalues <- vector(length=length(x), mode='numeric')
stat <- calculateStatistic(x)
for (i in 1:length(x)) {
jack <- x[-i]
jackStat <- calculateStatistic(jack)
pseudovalues[i] <- stat - (length(x)-1)*(jackStat-stat)
}Lưu ý: nhiều người cho rằng nên tránh các vòng lặp trong R bằng mọi giá. Những gì tiếp theo trong khối tiếp theo là một cách để tính toán các giá trị giả mà không có vòng lặp. Như thường lệ, giải pháp là sử dụng apply () hoặc một trong những họ hàng của nó. Ở đây, chúng tôi sử dụng sapply (), vì nó có thể được áp dụng cho một vectơ, trái ngược với apply (), hoạt động trên một ma trận. Chúng tôi cung cấp hàm sapply () với một hàm ẩn danh (không được đặt tên) như một hàm đóng (trong dấu ngoặc nhọn), áp dụng các thao tác đó từng hàng một trong x. Việc đóng cửa thu nhận y và ước tính từ môi trường của nó. Điều này khá nâng cao, vì vậy nếu vòng lặp có ý nghĩa hơn đối với bạn, thì hãy sử dụng nó.
y <- x
stat <- calculateStatistic(x)
pseudovalues <- sapply(x, function(x) {
i <- min(which(y==x))
jack <- y[-i]
jackStat <- calculateStatistic(jack)
thePseudovalue <- stat - (length(x)-1)*(jackStat - stat)
thePseudovalue
})Bất kể chúng tôi sử dụng cách tiếp cận nào để tính toán các giá trị giả, chúng tôi có thể tính ước tính, sai số chuẩn và giới hạn tin cậy của mình theo cùng một cách.
estimate <- mean(pseudovalues) n <- length(pseudovalues) SE <- sd(pseudovalues)/sqrt(n) # confidence limits alpha <- 0.05 lowerCL <- estimate + qt(p=alpha/2, df=n-1) * SE upperCL <- estimate - qt(p=alpha/2, df=n-1) * SE
Tóm lại, jackknife có ba bước chính:
- xóa một điểm dữ liệu,
- tính toán thống kê và giá trị giả
- lặp lại quá trình này, loại bỏ một điểm dữ liệu tại một thời điểm để xây dựng một tập hợp n giá trị giả sử dụng các giá trị giả để ước tính tham số và độ không đảm bảo
Kiểm định Permutation
Kiểm định Permutation là một phương pháp resampling mạnh mẽ dùng để kiểm tra giả thuyết thống kê, đặc biệt khi không rõ phân phối của dữ liệu. Phương pháp này hoạt động bằng cách tính toán tất cả các hoán vị có thể của dữ liệu giữa các nhóm và so sánh kết quả thống kê từ dữ liệu thực với phân phối của kết quả thống kê từ các hoán vị. Điều này cho phép xác định xem sự khác biệt giữa các nhóm có đáng kể hơn những gì có thể xảy ra do ngẫu nhiên hay không, mà không cần giả định về phân phối cụ thể của dữ liệu.
Kiểm Định Permutation Trong R
Trong R, bạn có thể thực hiện kiểm định Permutation sử dụng các hàm tùy chỉnh hoặc thông qua các gói như permute:
# Giả sử có hai nhóm dữ liệu A và B
data_A <- c(...)
data_B <- c(...)
# Hàm tính toán statistic, ví dụ: sự khác biệt về trung bình
calculate_statistic <- function(data1, data2) {
abs(mean(data1) - mean(data2))
}
# Kết quả thực từ dữ liệu
observed_statistic <- calculate_statistic(data_A, data_B)
# Thực hiện Permutation
permutation_statistic <- replicate(1000, {
combined <- sample(c(data_A, data_B))
permuted_A <- combined[1:length(data_A)]
permuted_B <- combined[(length(data_A)+1):(length(data_A)+length(data_B))]
calculate_statistic(permuted_A, permuted_B)
})
# Tính p-value
p_value <- mean(permutation_statistic >= observed_statistic)Kiểm Định Permutation Trong Python
Trong Python, bạn có thể sử dụng thư viện numpy để thực hiện các phép tính cần thiết cho kiểm định Permutation:
import numpy as np
# Dữ liệu của hai nhóm A và B
data_A = np.array([...])
data_B = np.array([...])
# Hàm tính statistic
def calculate_statistic(data1, data2):
return abs(np.mean(data1) - np.mean(data2))
# Kết quả thực từ dữ liệu
observed_statistic = calculate_statistic(data_A, data_B)
# Thực hiện Permutation
permutation_statistic = []
for _ in range(1000):
combined = np.random.permutation(np.concatenate((data_A, data_B)))
permuted_A = combined[:len(data_A)]
permuted_B = combined[len(data_A):]
statistic = calculate_statistic(permuted_A, permuted_B)
permutation_statistic.append(statistic)
# Tính p-value
p_value = np.mean(permutation_statistic >= observed_statistic)Ví Dụ Minh Họa
Giả sử bạn muốn so sánh hiệu suất của hai phương pháp đào tạo khác nhau đối với nhóm vận động viên. Sử dụng kiểm định Permutation, bạn có thể tính toán sự khác biệt về hiệu suất giữa hai nhóm sau khi đã áp dụng mỗi phương pháp đào tạo, sau đó so sánh kết quả này với phân phối của sự khác biệt dưới giả định không có sự khác biệt (giả thuyết null). Từ đó, bạn có thể quyết định liệu có bằng chứng đủ để bác bỏ giả thuyết null và kết luận rằng một phương pháp đào tạo cụ thể có hiệu quả hơn.
Một số lưu ý khi sử dụng Resampling
Khi áp dụng các phương pháp resampling như Bootstrap và Permutation Testing, có một số điểm cần lưu ý để đảm bảo rằng kết quả phân tích là chính xác và có ý nghĩa. Dưới đây là một số lưu ý quan trọng:
Tác Động của Kích Thước Mẫu
- Kích Thước Mẫu Nhỏ: Các phương pháp resampling có thể không đáng tin cậy với kích thước mẫu rất nhỏ vì phân phối mẫu của ước lượng có thể không được ước lượng chính xác. Điều này đặc biệt đúng với Bootstrap, nơi mẫu nhỏ có thể dẫn đến sự biến động lớn trong ước lượng.
- Mẫu Lớn: Với mẫu dữ liệu lớn, kiểm định Permutation có thể trở nên rất tốn kém về mặt tính toán vì số lượng hoán vị có thể tăng lên một cách nhanh chóng.
Số Lần Lặp
- Số lần lặp ảnh hưởng đến độ chính xác của ước lượng trong Bootstrap và p-value trong Permutation Testing. Một số lượng lặp lớn có thể cải thiện độ chính xác nhưng cũng tăng thời gian tính toán. Thông thường, một số lần lặp từ 1,000 đến 10,000 được xem là đủ cho hầu hết các ứng dụng.
Hạn Chế và Cảnh Báo
- Phụ Thuộc vào Dữ Liệu: Cả hai phương pháp đều phụ thuộc vào giả định rằng mẫu dữ liệu gốc là đại diện cho tổng thể. Nếu mẫu dữ liệu không đại diện, kết quả resampling có thể không hợp lệ.
- Giả Định về Độc Lập: Đối với Permutation Testing, giả định về sự độc lập giữa các quan sát là quan trọng. Nếu dữ liệu có cấu trúc phụ thuộc, kết quả kiểm định có thể bị sai lệch.
- Sử Dụng Cẩn Thận với Dữ Liệu Có Cấu Trúc Phức Tạp: Trong trường hợp dữ liệu có cấu trúc phức tạp hoặc có tương tác cao giữa các biến, việc áp dụng trực tiếp các kỹ thuật resampling mà không cân nhắc có thể dẫn đến kết quả sai lệch.
- Kiểm Tra Giả Định: Trước khi áp dụng resampling, cần kiểm tra các giả định liên quan đến dữ liệu và đảm bảo rằng phương pháp được chọn phù hợp với bản chất của dữ liệu.
Khi áp dụng resampling, nhà khoa học dữ liệu cần phải cân nhắc cẩn thận về mục tiêu của phân tích, bản chất của dữ liệu, và các giả định đằng sau phương pháp để đảm bảo rằng kết quả cuối cùng là hợp lệ và có ý nghĩa.


